
Se trata de un gran avance para la biología sintética. Para lograrlo, los investigadores diseñaron genéticamente distintos tipos de bacteria con distintas capacidades para solucionar el problema de forma conjunta. El enfoque podría cumplir los sueños más ambiciosos de este campo científico
La Escherichia coli (E. coli) crece en nuestras entrañas, a veces con un efecto desafortunado, pero también facilita avances científicos en genética, biocombustibles e incluso en la vacuna de Pfizer contra el coronavirus (COVID-19), por nombrar algunos. Y, por si fuera poco, esta bacteria de múltiples talentos acaba de mostrar uno nuevo: puede resolver un clásico problema de laberinto informático con computación distribuida, dividiendo los cálculos necesarios entre diferentes tipos de células modificadas genéticamente.
Esta sorprendente hazaña es un éxito de la biología sintética, que tiene como objetivo crear circuitos biológicos similares a los electrónicos y programar células con la misma facilidad que los ordenadores.
El experimento del laberinto es una parte de lo que algunos investigadores consideran una dirección prometedora para el campo: en vez de diseñar un único tipo de célula para que haga todo el trabajo, diseñan varias, cada una con diferentes funciones, para realizar la tarea. Al colaborar, estos microbios diseñados podrían ser capaces de "computar" y resolver problemas como redes multicelulares de la naturaleza.
Hasta ahora, para bien o para mal, los biólogos sintéticos no solían aprovechar el poder del diseño de la biología al completo y eso les frenaba. La bióloga sintética de la Universidad de Harvard (EE. UU.) Pamela Silver afirma: "La naturaleza puede hacer justo eso (por ejemplo, el cerebro), pero nosotros aún no sabemos diseñar a ese abrumador nivel de complejidad usando la biología".
Este estudio con la E. coli, dirigido por el biofísico del Instituto Saha de Física Nuclear en Calcuta (la India) Sangram Bagh, representa un problema simple y divertido para "jugar". Pero también sirve como prueba de principio para la computación distribuida entre células, demostrando que otros problemas computacionales más complejos y prácticos podrían resolverse de manera similar. Si este enfoque consigue funcionar a mayor escala, podría ofrecer nuevas aplicaciones relacionadas con casi todo, desde los productos farmacéuticos hasta la agricultura y los viajes espaciales.
El bioingeniero de la Universidad de Toronto (Canadá) David McMillen afirma: "A medida que avanzamos en la solución de problemas más complejos con sistemas biológicos diseñados, distribuir la carga de esta manera se convertirá en una capacidad importante".
Construir un laberinto de bacterias
Lograr que la E. coli resolviera el problema del laberinto requirió cierto ingenio. Las bacterias no deambulaban por un laberinto palaciego de setos bien podados. Más bien, analizaron varias configuraciones de laberintos. La organización fue: un laberinto por cada tubo de ensayo, cada uno generado por una mezcla química diferente.
Las recetas químicas se extrajeron de una cuadrícula de 2×2 que representa el problema del laberinto. El cuadrado superior izquierdo de la cuadrícula es el inicio del laberinto y el cuadrado inferior derecho es el destino. Cada cuadrado de la cuadrícula puede ser un camino abierto o bloqueado, lo que genera 16 posibles laberintos.
Bagh y sus colegas tradujeron matemáticamente este problema en una tabla compuesta por unos y ceros, que muestra todas las posibles configuraciones del laberinto. Luego mapearon esas configuraciones en 16 mezclas diferentes de cuatro productos químicos. La presencia o ausencia de cada sustancia química corresponde a si un cuadrado en particular está abierto o bloqueado en el laberinto.
El equipo diseñó varios conjuntos de E. coli con circuitos genéticos diferentes que detectaban y analizaban esos químicos. En su conjunto, la población mixta de bacterias funciona como la computación distribuida; cada uno de los distintos conjuntos de células realiza una parte del cálculo, procesando la información química y resolviendo el laberinto.
Para realizar el experimento, los investigadores primero introdujeron la E. coli en 16 tubos de ensayo, añadieron una mezcla diferente de laberinto químico en cada uno de ellos y dejaron que las bacterias crecieran. Después de 48 horas, si la E. coli no detectaba un camino claro a través del laberinto, es decir, si los químicos requeridos no estaban ahí, entonces el sistema permanecía oscuro. Si la combinación química correcta estaba presente, los circuitos correspondientes se "encendían" y las bacterias expresaban colectivamente las proteínas fluorescentes, en amarillo, rojo, azul o rosa, para indicar las soluciones. "Si hay un camino, una solución, las bacterias brillan", explica Bagh.
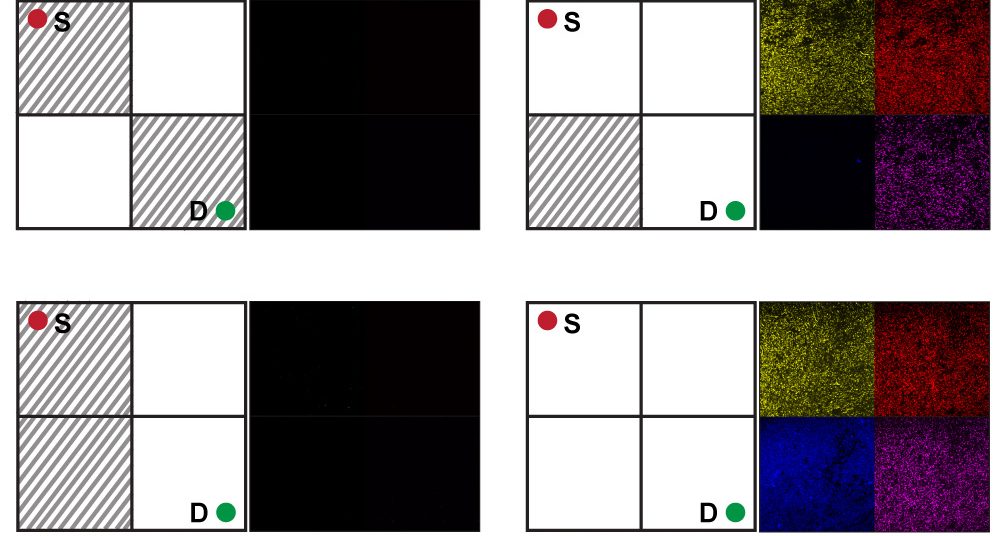
Foto: Cuatro de las 16 configuraciones posibles del laberinto. Los dos laberintos de la izquierda no tienen caminos despejados desde el inicio hasta el destino (debido a los cuadrados obstruidos); por lo tanto, no hay una solución y el sistema está oscuro. Para los dos laberintos de la derecha, hay caminos claros (cuadrados blancos), por lo que la E. coli que soluciona los laberintos brilla: las bacterias expresan colectivamente las proteínas fluorescentes, indicando así las soluciones. Créditos: Kathakali Sarkar y Sangram Bagh
Lo que Bagh encontró especialmente fascinante fue que, al recorrer los 16 laberintos, la E. coli proporcionó una prueba física de que solo tres se podían resolver. "Calcular esto con una ecuación matemática no es sencillo. Con este experimento, es posible visualizarlo de manera muy simple", explica.
Grandes objetivos
Bagh cree que un ordenador biológico de este tipo ayudará en criptografía y esteganografía (el arte y la ciencia de ocultar información), que utilizan los laberintos para cifrar y ocultar datos, respectivamente. Pero las implicaciones se van mucho más allá de esas aplicaciones para cumplir las ambiciones más elevadas de la biología sintética.
La idea de la biología sintética se remonta a la década de 1960, pero el campo surgió concretamente en el año 2000 con la creación de los circuitos biológicos sintéticos (específicamente, un interruptor de palanca y un oscilador) que permitieron programar las células para producir compuestos deseados o reaccionar de forma dirigida dentro de sus entornos.
La biología, sin embargo, no ha sido el colaborador más cooperativo. Uno de sus factores limitantes es la cantidad de cambios que se pueden realizar en una célula sin destruir su viabilidad. "La célula tiene sus propios intereses", destaca McMillen, cuyo laboratorio está desarrollando un sistema basado en la levadura que detecta los anticuerpos contra la malaria en las muestras de sangre y un sistema similar para la COVID-19. Al insertar componentes diseñados por humanos en un sistema biológico, McMillen señala que se "lucha contra la selección natural y la entropía, que son dos de las grandes fuerzas de la naturaleza".
Si una célula está sobrecargada de extras, por ejemplo, existe el riesgo de interferencia y diafonía, lo que obstaculiza el rendimiento y limita las capacidades del sistema. En cuanto a los laberintos computacionales, Bagh explica que el algoritmo podía haberse programado con un solo tipo de célula de E. coli. Pero funcionó mejor cuando las funciones necesarias se distribuyeron entre seis tipos de células.
"Existe un límite físico sobre la cantidad de partes genéticas que se pueden usar en una sola célula. Esto limita la sofisticación de los conceptos informáticos que se pueden desarrollar", afirma la ingeniera química del Imperial College de Londres (Reino Unido) Karen Polizzi, que desarrolla biosensores celulares para monitorizar la fabricación de proteínas terapéuticas y vacunas.
"La computación distribuida podría ser una manera de conseguir algunos de los objetivos más elevados de la biología sintética, porque no hay forma de que una célula realice una tarea compleja por sí sola", añade.
Supremacía celular
El biólogo sintético del MIT y editor en jefe de ACS Synthetic Biology (donde se publicó el resultado de Bagh), Chris Voigt, cree que la computación distribuida es la dirección que debe tomar la biología sintética.
En su opinión, tener grandes ambiciones para las células microbianas tiene sentido, y él mismo ha realizado cálculos para respaldarlas. Y afirma: "Creo que una cucharadita de bacterias tiene más puertas lógicas que 2.000 millones de procesadores Xeon. Y tiene más memoria en el ADN que todo internet. La biología tiene una capacidad de cálculo increíble y lo hace distribuyéndola en una enorme cantidad de células". Pero hay salvedades: "Una puerta tarda 20 minutos en procesarse, por lo que es un proceso muy lento", matiza.
El año pasado, Voigt y sus colaboradores lograron programar el ADN con un algoritmo de calculadora y generar una pantalla digital con E. coli fluorescente. Utilizaron un software creado por el laboratorio de Voigt, llamado Cello, que toma los archivos de Verilog, el lenguaje de programación utilizado para describir y modelar circuitos, y los convierte en ADN, por lo que se pueden ejecutar los mismos tipos de configuraciones en las células. Sin embargo, todos los circuitos de la calculadora de E. coli estaban agrupados en un solo tipo de célula, en una sola colonia. Ante eso, admite: "Hemos llegado al límite de ese enfoque. Tenemos que descubrir cómo hacer diseños más grandes".
Incluso si los investigadores quisieran usar bacterias para ejecutar algo muy sencillo para los estándares actuales, como los sistemas de guía del Apolo 11, Voigt asegura que no se podría hacer con una sola célula diseñada. Y añade: "La capacidad está ahí. Solo necesitamos formas de dividir el algoritmo en las células y luego vincularlas para que compartan la información de manera eficiente y puedan realizar el cálculo de manera colectiva".
De hecho, se pregunta si imitar la computación electrónica tradicional es el mejor enfoque para aprovechar el poder computacional de la biología y resolver los problemas complejos de base biológica.
En su búsqueda del enfoque más adecuado, Bagh también ideó un tipo de arquitectura de red neuronal artificial para el hardware bacteriano. Además, está interesado en explorar un enfoque que involucre la "lógica difusa", yendo más allá de las limitaciones de los ceros y unos binarios hacia un continuo más alineado con el ruido y el desorden de los sistemas biológicos vivos.
El biólogo sintético de la Universidad Politécnica de Madrid (España) Ángel Goñi-Moreno coindice: "Si vamos a jugar con tecnología viva, tenemos que seguir las reglas de los sistemas vivos".
Plantea romper con la analogía del circuito electrónico capitalizando cómo las células perciben, responden y se adaptan a su entorno, utilizando la propia selección natural como una herramienta para impulsar los diseños computacionales. La evolución, según él, es un proceso biológico que computa la información a lo largo del tiempo, optimizando los sistemas celulares para lograr una diversidad de tareas.
El español cree que este enfoque podría culminar en lo que denomina como "supremacía celular". El término plantea un paralelismo deliberado con la "supremacía cuántica" (últimamente denominada más como "primacía cuántica"), el punto en el que los ordenadores cuánticos superan las capacidades de la computación convencional en ciertos campos. Los bioordenadores que evolucionen hasta tal punto podrían ofrecer, en palabras de Goñi-Moreno, una mayor habilidad para resolver problemas en áreas como la mejora de la producción agrícola (como las bacterias del suelo que pueden ajustar los productos químicos que producen en función de las condiciones cambiantes) y la terapéutica de enfermedades.
Pero no espere que la E. coli le ayude a navegar por internet o resuelva el problema de P vs. NP; para eso, todavía necesitaremos buenos ordenadores convencionales.


